R语言学习笔记7
R语言教程-R编程4
18 R程序效率
R的运行效率
R是解释型语言,在执行单个运算时, 效率与编译代码相近; 在执行迭代循环时, 效率较低, 与编译代码的速度可能相差几十倍。 在循环中对变量进行修改尤其低效。
R以向量、矩阵为基础运算单元, 在进行向量、矩阵运算时效率很高, 应尽量采用向量化编程。
R语言的设计为了方便进行数据分析和统计建模, 有意地使语言特别灵活, 比如, 变量为动态类型而且内容可修改, 变量查找在当前作用域查找不到可以向上层以及扩展包中查找, 函数调用时自变量仅在使用其值时才求值(懒惰求值), 这样的设计都为运行带来了额外的负担, 使得运行变慢。
在计算总和、元素乘积或者每个向量元素的函数变换时, 应使用相应的函数,如sum, prod, sqrt, log等。
为了提高R程序的运行效率, 需要尽可能利用R的向量化特点, 尽可能使用已有的高效函数, 还可以把运行速度瓶颈部分改用C++等编译语言实现, 可以用R的profiler工具查找运行瓶颈。 对于大量数据的长时间计算, 可以借助于现代的并行计算工具。
对已有的程序, 仅在运行速度不满意时才需要进行改进, 否则没必要花费宝贵的时间用来节省几秒钟的计算机运行时间。 要改善运行速度, 首先要找到运行的瓶颈, 这可以用专门的性能分析(profiling)功能实现。 R软件中的Rprof()函数可以执行性能分析的数据收集工作, 收集到的性能数据用summaryRprof()函数可以显示运行最慢的函数。 在RStudio中,可以用Profile菜单执行性能数据收集与分析, 可以在图形界面中显示程序中哪些部分运行花费时间最多。
如果要实现一个比较单纯的不需要利用R已有功能的算法, 发现用R计算速度很慢的时候, 也可以考虑先用Julia语言实现。 Julia语言设计比R更先进,运算速度快得多, 运算速度经常能与编译代码相比, 缺点是刚刚诞生几年的时间, 可用的软件包还比较少。
向量化编程
计算: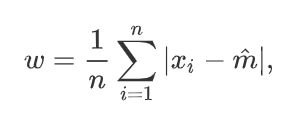
其中x1,x2,…xn是某总体的样本,m head 是样本中位数。 用传统的编程风格, 把这个统计量的计算变成一个R函数,可能会写成:
f1 <- function(x){ |
用R的向量化编程,函数体只需要一个表达式:
f2 <- function(x) mean(abs(x-median(x))) |
其中x - median(x)利用了向量与标量运算结果是向量每个元素与标量运算的规则, abs(x - median(x))利用了abs()这样的一元函数如果以向量为输入就输出每个元素的函数值组成的向量的规则,mean(…)避免了求和再除以n的循环也不需要定义多余的变量n。
median(c(2,4)) |
在R中, 用system.time()函数可以求某个表达式的计算时间, 返回结果的第3项是流逝时间。比较两个程序的效率:这步需要一点时间才能运行完成
# 产生10000个随机数 |
我的电脑运行结果显示时间相差5.2倍
17.49/3.36 |
有一个R扩展包microbenchmark可以用来测量比较两个表达式的运行时间。
install.packages('microbenchmark') |
示例2
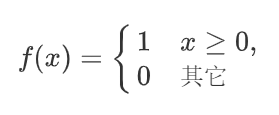
一般程序写成:
f1 <- function(x){ |
也可以写为:
f2 <- function(x){ |
利用R中内建函数ifelse(), 可以把函数体压缩到仅用一个语句:
f3 <- function(x) ifelse(x >= 0, 1, 0) |
示例3

普通循环写成:
f1 <- function(){ |
用prod()函数可以向量化内层循环:
f2 <- function(){ |
把程序用cumprod()函数改写, 可以完全避免循环:
f3 <- function(){ |
sum,prod:向量元素和,积 cumsum,cumprod:累加、累乘
# 返回2*3*4*5的积 |
用microbenchmark比较速度:
microbenchmark::microbenchmark( |
f3()比f1()快2千多倍
减少显示循环
显式循环是R运行速度较慢的部分, 有循环的程序也比较冗长, 与R的向量化简洁风格不太匹配。 另外, 在循环内修改数据子集,例如数据框子集, 可能会先制作副本再修改, 这当然会损失很多效率。 R 3.1.0版本以后列表元素在修改时不制作副本, 但数据框还会制作副本。
R中的有些运算可以用内建函数完成, 如sum, prod, cumsum, cumprod, mean, var, sd等。 这些函数以编译程序的速度运行, 不存在效率损失。
replicate()函数
replicate()函数用来执行某段程序若干次, 类似于for()循环但是没有计数变量。 常用于随机模拟。
语法为:
replicate(重复次数, 要重复的表达式)
其中的表达式可以是复合语句, 也可以是执行一次模拟的函数。
其中的表达式可以是复合语句, 也可以是执行一次模拟的函数。
set.seed()用于设定随机数种子,让产生的随机数能够再次出现
set.seed( ) 括号的数只是一个编号,作为标记使用,取值可以随意
当以后需要取得与上次相同的随机数时, set.seed( ) 中填写回上面设置的值即可。
例子:
set.seed(1) |
避免制作副本
类似于x <- c(x, y), x <- rbind(x, y)这样的累积结果每次运行都会制作一个x的副本, 在x存储量较大或者重复修改次数很多时会减慢程序。
set.seed(101) |
上面的程序不仅是低效率的做法, 也没有预先精心设计用来保存结果的数据结构。 数据建模或随机模拟的程序都应该事先分配好用来保存结果的数据结构,在每次循环中填入相应结果。
set.seed(333) |
在循环内修改数据框的值也会制作数据框副本, 当数据框很大或者循环次数很多时会使得程序很慢。如:
set.seed(2) |
在循环内修改列表/矩阵元素就不会制作副本, 从而可以避免这样的效率问题,如:
set.seed(101) |
也可以写为:
set.seed(101) |
replicate()函数中用simplify=FALSE使结果总是返回列表。 要注意的是, 最后数据转为数据框后也是效率较差的。 将数据保存在列表中比保存在数据框中访问效率高, 数据框提供的功能更丰富。
R的计算函数
R中提供了大量的数学函数、统计函数和特殊函数, 可以打开R的HTML帮助页面, 进入“Search Enging & Keywords”链接, 查看其中与算术、数学、优化、线性代数等有关的专题。
例举几个函数:
sum对向量求和, prod求乘积。
cumsum和cumprod计算累计, 得到和输入等长的向量结果。
diff计算前后两项的差分(后一项减去前一项)。
mean计算均值,var计算样本方差或协方差矩阵, sd计算样本标准差, median计算中位数, quantile计算样本分位数。 cor计算相关系数。
colSums, colMeans, rowSums, rowMeans对矩阵的每列或每行计算总和或者平均值, 效率比用apply函数要高
max和min求最大和最小, cummax和cummin累进计算。
range返回最小值和最大值两个元素。
pmax(x1,x2,…)对若干个等长向量计算对应元素的最大值, 不等长时短的被重复使用。 pmin类似。 比如,pmax(0, pmin(1,x))把x限制到内。
排序
sort返回排序结果。 可以用decreasing=TRUE选项进行降序排序。 sort可以有一个partial=选项, 这样只保证结果中partial=指定的下标位置是正确的。 比如:
# 第三个数的排序是正确的 |
在sort()中用选项na.last指定缺失值的处理, 取NA则删去缺失值, 取TRUE则把缺失值排在最后面, 取FALSE则把缺失值排在最前面。
order, sort.list, rank也可以有 na.last选项,只能为TRUE或FALSE。
unique()返回去掉重复元素的结果, duplicated()对每个元素用一个逻辑值表示是否与前面某个元素重复。
unique(c(1,2,2,3,1)) |
一元函数求根uniroot
uniroot(f, interval)对函数f在给定区间内求一个根, interval为区间的两个端点。 要求f在两个区间端点的值异号。 即使有多个根也只能给出一个。
uniroot(function(x) x*(x-1), c(1,4)) |


